前の記事(
1、2 )で、生物学的実験に応じてデータがどのように見えるかを知りました。 これらの視覚化されたデータに基づいて、セル内で何が起こっているかについての仮定が行われました。 次に、機械がルーチンを実行できるように、データを数学的およびアルゴリズム的に分析する方法について説明します。 残念ながら、データ分析に関する多くの記事を読んだ後、単一または最も普遍的なソリューションはないという印象を受けました。 特定のデータセットでうまく機能するアルゴリズムがあり、他のケースではタスクを満たしていない場合があります。
まず、この記事で使用した基本的な概念を少し思い出して説明したいと思います。 edとその入手方法について話すたびに、驚きと誤解に遭遇します。 そして、たまたま、紙の束をシュレッダーに投げて、私はあなたがどのように読んだかを説明しようとすることができることに気付きました。 本があり、本自体がセルであり、本の各章がDNAであるとします。 この本の数百万のコピーを在庫しています。 あなたは本をシュレッダーに送り、シュレッダーはそれらを細断しました。 ここでは、細断された紙片の入った容器を用意し、それらをよく混ぜて最初に手に入れたものを手に入れます。 次に、ストリップを慎重にレイアウトし、両側から小片を切り取り、切り取った部分を別の箱に折ります。 これらの小さな破片-とリード、ストライプがあります-これらは断片です。 次に、特定のプログラムの助けを借りて、カットされたピースが本のどの部分にあるかを判断します。 幸運にも時々、本の中で作品が切り取られた場所を特定できます。 たとえば、一部の章が前の章を繰り返すことから始まった、またはすべての操作の結果として、一部の文字が消去されて本に見つからなかったという事実のために、プログラムはこの作品の場所にいくつかの可能なオプションを決定することがあります。 ストライプの断片で何が起こるかというトピックに関するバリエーションの数は非常に多く、それらすべてについて議論すると、アルゴリズムに到達できません。 何が何であるかがより明確になることを願っています
アルゴリズムを説明し、開発者がテストしたデータを使用してそれについて説明します。 しかし、これは別の種類のデータに使用できないことを意味するものではありません。 また、私が遭遇したアルゴリズムの欠点を特定しようとします。 アルゴリズムを体系化するつもりはありませんが、シーケンスされたデータの一般的な分析で使用された順序でグループに分けて説明します。 この記事では、「アダプターによる汚染」の簡単な分析と、景観の予備的な準備について、そしておそらく次の記事で、または取得したデータの品質の評価について説明します。
アダプターの汚染
シーケンス機器会社も、関連キット(スライド、チューブ、試薬)を製造しています。 試薬の1つのタイプは、アダプターと呼ばれる配列決定に必要なポリヌクレオチド鎖です。
準備された材料にはさまざまな種類の汚染があります。 それらの1つはアダプター汚染です。 このような汚染の分析は簡単です。 1番目、2番目などのヌクレオチドの頻度を計算する必要があります。 受信したFASTA / FASTQファイルの位置。 得られたデータに基づいて、テーブルがコンパイルされます。 次の例を使用して、分析プロセスをより詳細に検討してください。表の最初の9列を以下に示します(表1を参照)。
表1
| POS / NUCL | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| A | 446500 | 2869138 | 599400 | 941816 | 1145756 | 1133404 | 3599581 | 3291736 |
| C | 1922795 | 1367021 | 1761361 | 3723679 | 1030494 | 1173573 | 963827 | 1284534 |
| T | 485441 | 1337742 | 3273475 | 918001 | 1016981 | 1006168 | 1494250 | 1291715 |
| G | 4130093 | 1425977 | 1365764 | 1416504 | 3806769 | 3686855 | 942342 | 1132015 |
| N | 15171 | 122 | | | | | | |
表のデータを使用して、グラフを作成できます(図1)。 グラフのピークは、現在の位置で最もよく見られる文字(ヌクレオチド)に対応しています。 特に、最初の文字「G」、2番目の文字「A」、3番目の位置の「T」。
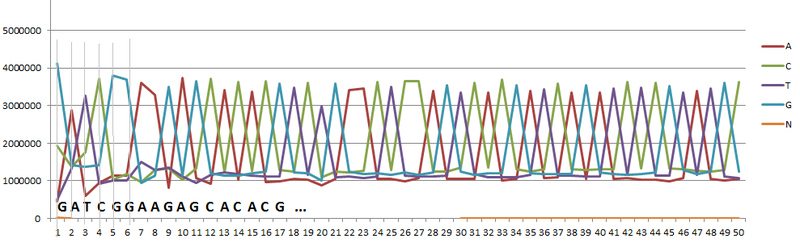
図 1
ピークに対応する文字のチェーン全体を構成すると、ソースデータで最もよく見られるシーケンスが得られます。 たとえば、検討中の例のように、このシーケンスがアダプターのリストのシーケンス(シーケンス用の機器の製造元から提供されている)と一致した場合、イルミナのアダプターシーケンスの1つが取得され、アダプターが汚染されています。
アダプターと原料の汚染の割合を計算するには、次の簡単な手順を実行します。 1.表のいずれかの位置にある文字の総数を数えます。この例では正確に7000000です。2.汚染がなかった場合、各位置の文字の数はほぼ同じです。 ピークに対応する文字を考慮から除外して、残りの文字を考慮します。 グラフから次のように、各位置に約120万の文字があります3.各位置に同じ数の文字があるはずなので、1,200,000 * 4 = 4,800,000になります。4.したがって、アダプターの汚染は4,800,000 / 7,000,000 *です。 100%= 32%。 つまり 次に材料を準備するときに、追加するアダプタの数を減らす必要があります。
後で分析するためのプロファイルの準備
私が知っているアルゴリズムのほとんどは、初期データ処理を実行します-プロファイルを準備します。 つまり、データをグループ化し、その後、重要度レベルを評価します。 グループ化されたデータは、DNA上のタンパク質の可能性のある場所の位置を決定します。これは、アルゴリズムを使用して見つけようとしています。
クロマチン免疫沈降の使用の結果として得られたデータを考慮してください(より詳細には、
DNAシーケンス )。 この方法の特徴を思い出させてください。フラグメントは、目的のタンパク質に対応する抗体を使用して選択されるため、ほとんどのフラグメントにはタンパク質が結合した配列が含まれています。 また、スライスすると、さまざまなサイズのフラグメントが得られます;さらに、長さに沿ってフィルタリングされます(150〜300ヌクレオチド以下)。 最終結果のほとんどの読み取りは、タンパク質の両側にあります。 将来この機能を使用して、タンパク質の推定位置を決定します。
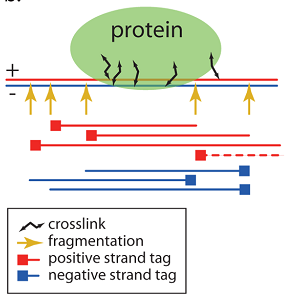
図 2
図2では、黄色の矢印はDNA切開の可能性のある部位を示しています;ここでフラグメントが開始または終了できます。 赤い線は正のスパイラル(図では「+」記号で表示)から取得した読み取り値を示し、青い線は負の値(「-」記号で表示)を示します。 場合によっては、より高価な実験(「
ペアエンドシーケンス 」)を実行すると、シーケンス装置は1つのフラグメントから取得した2つのリードを判別できます。 この方法を数回使用すると、タンパク質の推定位置を決定する品質が向上することも主張されています[1]。 ペアエンド方式はまだあまり使用されないため、ほとんどの場合、無関係な一連の読み取りを処理します。
理想的なケースでは、図3に示すように、タンパク質の周囲の読み取り位置のランドスケープがあります。実験のランダム成分の数が多い場合、これはまれです。

図 3
以下は、実験データからの風景の例です(図4)。 列(ウィンドウ)の幅は20ヌクレオチドに対応し、列の最大高さは5〜8リードのみです。 ウィンドウの幅が1ヌクレオチドに等しい場合、画像はさらに離散的でノイズが多くなります。 ただし、以下で提案するプロファイルを準備するためのアルゴリズムによってデータが処理される場合、ランドスケープは理想にさらに近くなります(図3)。

図 4
手動プロファイルの準備
マシンのデータをより読みやすい形式にする方法は? 場合によっては、フラグメントが予想される長さを事前に知っています。 たとえば、H3K4Me3(MNaseメソッド)の場合、約148bpです[2]。 読み取り値をフラグメントの長さの半分だけシフトします。148/ 2 =74。正のスパイラルで見つかった読み取り値は右に74シフトし、負のスパイラルで見つかった値は左に74シフトします。絶対値を加算してグラフに表示します(図5)[3、 4]。
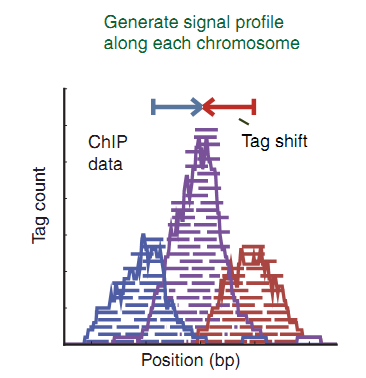
図 5
図(図5)では、紫の色は、スパイラルのプラス側とマイナス側からの読み取り回数を加算した結果のピークを示しています。 ピークピークは、ある程度の精度で、DNA上のタンパク質の中心に対応している必要があります。 このような単純な変換により、信号が増幅され、今ではより顕著になります。
上記のグラフをプロットするにはさまざまな方法があります。 1つの方法:「Y」軸で、「X」点からの読み取り回数を延期します。 別の方法:セグメントを使用して、「Y」軸に沿ってプロットし、特定のポイント「X」でのリッジの交点の数を延期します。 グラフが作成され、「Y」軸に沿って尾根ではなくフラグメントの交差が延期されることがあります。 このようなグラフの意味は、読み取り/フラグメントによるDNAカバレッジの密度です。
フラグメントのサイズを事前に見積もり、その値をプログラムに転送できると便利です。 しかし、フラグメントの長さがわからない場合、どうすればデータをシフトする量を計算できますか?
相互相関プロファイルの準備
シフトの大きさを計算する1つの方法は、正と負のスパイラルからの読み取りデータを相互相関させることです。 各染色体「c」に対して、関数を定義します
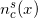
、各ポイント「x」で、スパイラル「s」側の読み取り回数に等しい値を取ります。 シフトの定義
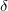
機能
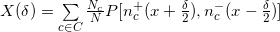
ここで:
 -ベクトル「a」と「b」間の線形ピアソン係数。
-ベクトル「a」と「b」間の線形ピアソン係数。- Cはすべての染色体のセットです。
- N cは染色体「c」の読み取り数です。
- Nは読み取りの総数です。
たとえば、シフト値が-100から300までの範囲を選択し、グラフを作成します(図6)[4]。
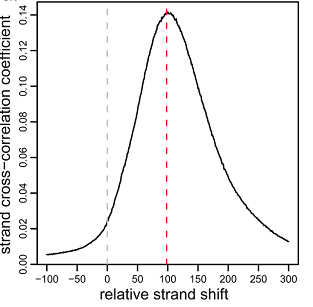
図 6
x軸に沿ったこのグラフのピークは、最大の相関を持つシフトに対応します。 図6に示す場合、シフトは100/2 = 50ヌクレオチドです。 すべての染色体を考慮したデータの相関は、関心領域の相関に比べて精度が低い場合があります。 だからあなたの
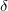
各強化セグメントのせん断。
カーネル密度プロファイル
カーネル密度関数[4,5,6]を使用するアルゴリズムを検討してください。 式に従って、各位置iのコーティング密度を再定義します
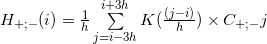
ここで:
- h-カーネル密度帯域幅、記事[5]では30bpが使用され、記事[4]では、図6に示すように、中央で0.45のピーク幅が使用されました。
 -ガウスカーネル密度関数。
-ガウスカーネル密度関数。 -正のスパイラルと負のスパイラルのポイントjで終わる読み取りの数をそれぞれ返します。
-正のスパイラルと負のスパイラルのポイントjで終わる読み取りの数をそれぞれ返します。
したがって、各スパイラルの密度を再計算し、信号を増幅すると、十分に濃縮されたセクションでは、スパイラルの各側のピークを決定し、シフト量についてそれらの差の半分を取ることができます(図7、「ピークシフト」)。
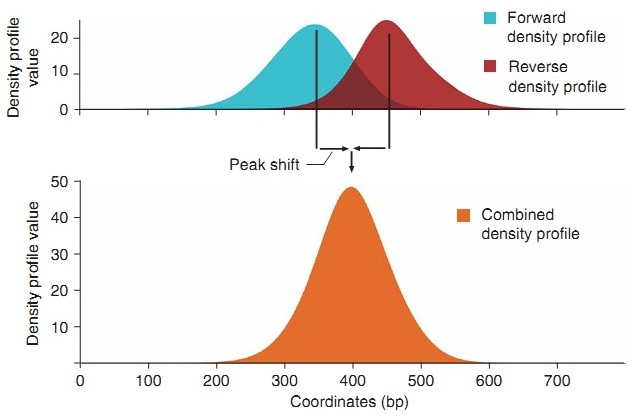
図 7
幾何学的プロファイル構築方法
このアルゴリズムは、単純な数学演算を使用します。 幅wのウィンドウと位置iに対して、p1(i)iの左側の正の螺旋上のウィンドウ内のタグの数として定義します。 p2(i)位置iの右側にあるタグの数。 負のスパイラルに対してのみ、pの定義に従ってn1(i)とn2(i)を定義します(図8.1)。 次の式を使用して、ポイントiの値を計算します。
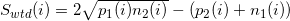
指定されたウィンドウ内で最大値を見つけます。 見つかった最大値は、推定タンパク質結合位置です。 このメソッドは、Windowsタグ密度からWTDと呼ばれていました。
記事[4]では、修正することも提案されました。

ここで、P [a、b]はベクトルaとbの間の線形ピアソン相関係数であり、ベクトル「a」は位置iからi + kまでの正のスパイラルの読み取り数であり、ベクトル「b」は数位置iからikまでの負のスパイラルを読み取り、

(図8.2)。 彼らはMirrow Tag CorrelationからMTCを呼び出しました。
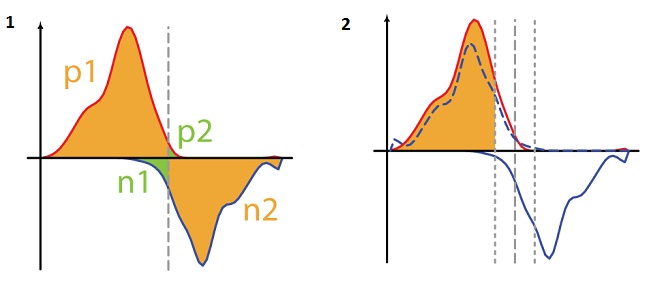
図 8
プロファイルの構築に使用される他のアルゴリズムがあります[7]。 最も一般的に使用されているものを強調してみました。 すべてのアルゴリズムは既にいくつかのソフトウェアパッケージに実装されており、リンクは記事に記載されています。その一部を次に示します。
sissrs.rajajothi.comhttp://home.gwu.edu/~wpeng/Software.htmmendel.stanford.edu/sidowlab/downloads/questhttp://compbio.med.harvard.edu/software.htmlアルゴリズムを使用したプロファイルの正しい構築は、すべてのプログラムの最も重要な部分です。プロファイルがより正確に構築されるほど、タンパク質の位置を特定する可能性が高くなるためです。 アルゴリズムの別の部分は、見つかったセクションの重要性を数学的に評価することを目的としています。 しかし、残念なことに、すべてのアルゴリズムは人を置き換えることはできませんが、データを日常的に処理するのに役立ち、検索を行うべき場所を暗示します。 そのため、生物学、化学、数学、プログラミングの相互作用のシステム全体は完全とはほど遠いものであり、アイデアの検索、実装、改善のための巨大な分野があります。
1. Wang、C.、et al。、ペアエンドChIP-Seqデータからのin vivoタンパク質-DNA結合部位の同定のための効果的なアプローチ。 BMC Bioinformatics、2010.11:p。 81.
www.ncbi.nlm.nih.gov/pmc/articles/PMC28318492. Barski、A.、et al。、ヒトゲノムにおけるヒストンメチル化の高解像度プロファイリング。 Cell、2007.129(4):p。 823-37。
www.ncbi.nlm.nih.gov/pubmed/175124143. Pepke、S.、B。Wold、およびA. Mortazavi、ChIP-seqおよびRNA-seq研究用の計算。 Nat Methods、2009.6(11 Suppl):p。 S22-32。
www.ncbi.nlm.nih.gov/pubmed/198442284. Kharchenko、PV、MY Tolstorukov、PJ Park、DNA結合タンパク質のChIP-seq実験の設計と分析。 Nat Biotechnol、2008.26(12):p。 1351-9。
www.ncbi.nlm.nih.gov/pubmed/190299155. Valouev、A.、et al。、ChIP-Seqデータに基づく転写因子結合部位のゲノムワイド分析。 Nat Methods、2008.5(9):p。 829-34。
www.ncbi.nlm.nih.gov/pubmed/191605186. Boyle、AP、et al。、F-Seq:高スループットシーケンスタグの特徴密度推定量。 バイオインフォマティクス、2008.24(21):p。 2537-8。
www.ncbi.nlm.nih.gov/pmc/articles/PMC27322847. Zang、C.、et al。、ヒストン修飾ChIP-Seqデータから濃縮ドメインを識別するためのクラスタリングアプローチ。 バイオインフォマティクス、2009.25(15):p。 1952-8。
いくつかの図面はnature.comから取られ、許可を得て印刷されました。
ライセンス日:2012年2月16日、
ライセンス番号:2850830942235、2850831421638
レビューは、ケンタッキー州コビントンのアンドレイ・カルタショフによって作成されました。porter@ porter.st