Rのforループは、特に大きなデータセットを処理する場合に、最適化せずに純粋な形式で適用すると非常に遅くなる可能性があります。 コードを高速化する方法はいくつかありますが、その量を知ると驚くでしょう。
この記事では、ロジックの単純な変更、並列処理、
Rcppを含むいくつかのアプローチについて説明します。速度を数桁向上させることで、1億行以上のデータを処理できるようになります。
forループと条件ステートメント(if-else)を使用してコードを高速化し、データセット(データフレーム、df)に追加される列を作成してみましょう。 以下のコードは、この初期データセットを作成します。
この部分では、ベクトル化、真の条件のみ、ifelse。
次の部分:which、apply、byte compilation、Rcpp、data.table。
最適化するロジック
このデータセット(df)の各行について、値の合計が4を超えるかどうかを確認します。4を超える場合、新しい5番目の変数は値 "greater_than_4"を取得し、そうでない場合は "lesser_than_4"を取得します。
それ以降のすべての処理時間の計算は、2.6 GHzプロセッサと8 GBのRAMを搭載したMAC OS Xで実行されました。
事前にデータ構造をベクトル化し、強調表示する
計算サイクルを開始する前に、必要な長さとデータ型を指定して、データ構造と出力変数を常に初期化します。 ループ内で段階的にデータ量を増やしないようにしてください。 1,000行から100,000行までのさまざまなデータサイズで、ベクトル化によって速度がどのように向上するかを比較してみましょう。
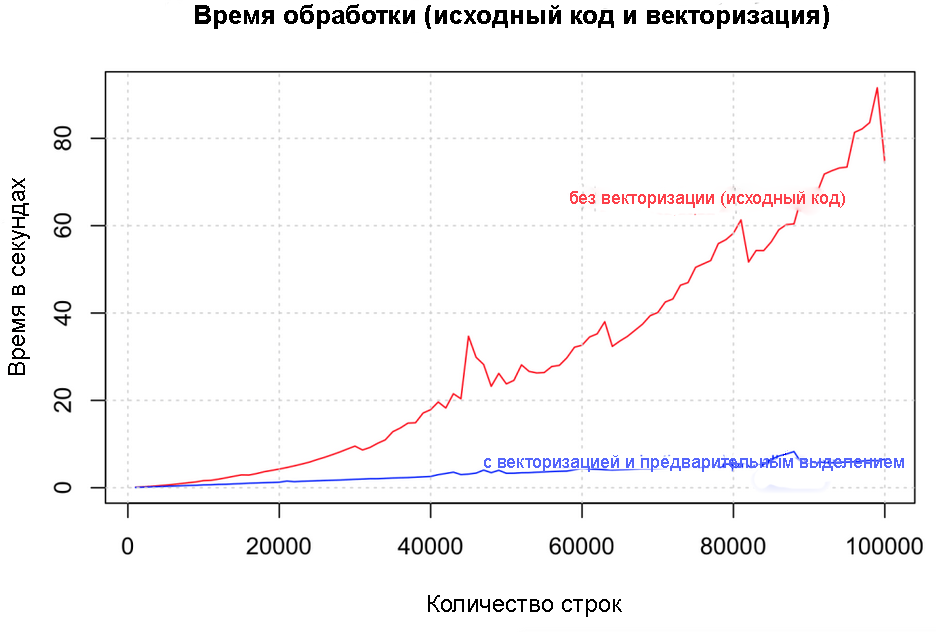 ソースコードとベクトル化されたコード
ソースコードとベクトル化されたコードループ外の条件ステートメントを削除する
条件付きチェックのサイクルから抜け出すことは、ゲインに関してはベクトル化自体と同等です。 テストは、100,000〜1,000,000行の範囲で実施されました。 速度の向上は再び巨大です。
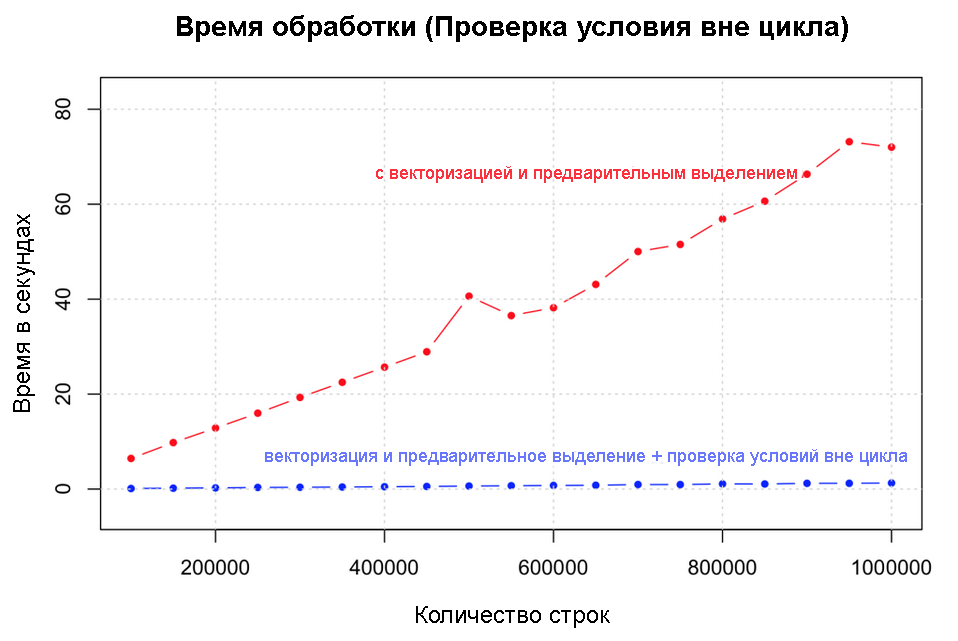 サイクル外の状態を確認する
サイクル外の状態を確認する真の条件に対してのみループを実行する
ここで使用できるもう1つの最適化は、出力ベクトルをFalse値で初期化した後、真の条件下でのみサイクルを開始することです。 ここでの加速は、データのTrueケースの数に大きく依存します。
テストでは、このパフォーマンスと、1,000,000行から10,000,000行のデータの以前の改善を比較します。 ここでゼロの数の増加に注意してください。 予想どおり、非常に明確な顕著な改善があります。
output <- character(nrow(df)) condition <- (df$col1 + df$col2 + df$col3 + df$col4) > 4 system.time({ for (i in (1:nrow(df))[condition]) {
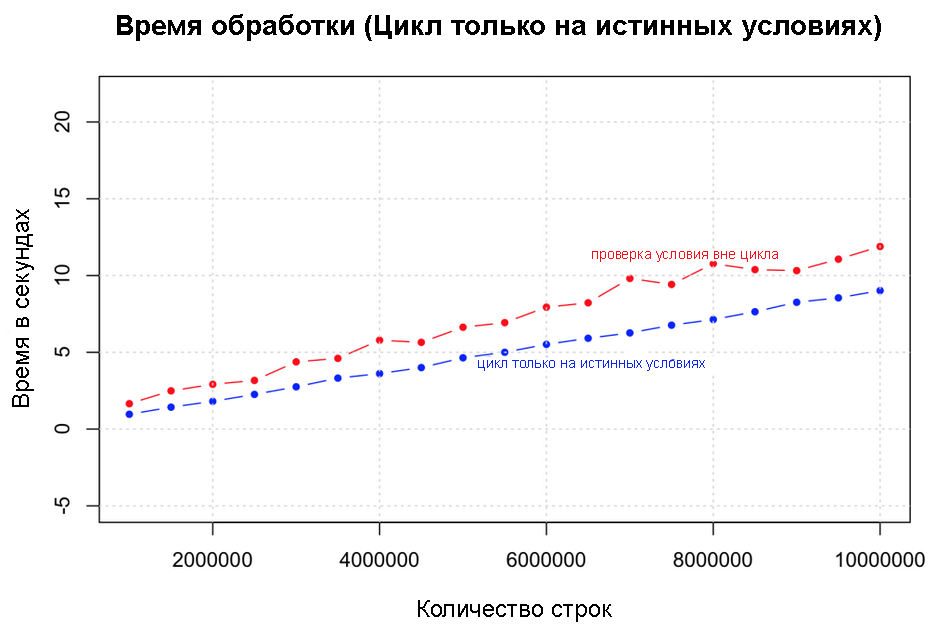 真の条件下でのみサイクルを開始する
真の条件下でのみサイクルを開始する可能な場合はifelse()を使用します
ifelse()を使用すると、このロジックをはるかに高速かつ簡単に作成できます。 構文はMS Excelの
if関数に似ていますが、特に予備選択がないことを考慮すると、加速は驚異的で、毎回条件がチェックされます。 これは、単純なループを高速化する非常に有益な方法のようです。
system.time({ output <- ifelse ((df$col1 + df$col2 + df$col3 + df$col4) > 4, "greater_than_4", "lesser_than_4") df$output <- output })
 真の条件とifelseのみ
真の条件とifelseのみ